这篇blog介绍2025年五月发表在nature computational science上的一篇论文,Quantifying batch effects for individual genes in single-cell data.
单细胞的批次效应是一个老生常谈的问题,也有大批有效的批次矫正方法。但少有工作尝试阐述问题,“批次效应到底是怎样影响数据的”。这篇工作给了一个很好的回答,特征可以通过简单的指标被划分为 batch-sensitive 和 batch-insensitive 两大类。使用batch-insensitive特征来做PCA就能很好的去除批次效应。
批次效应是怎样影响数据的?
批次效应体现在数据聚类中,不同来源的同种细胞会被划分到不同的cluster中(直观的体现在UMAP中,细胞根据批次来源聚在一起而不是根据细胞类型聚在一次)。一个显然的解释就是distribution drift. 那么这种分布怎样在特征上体现呢?
group technical effects(GTE)
作者引入一个指标GTE,量化一个特征在不同批次间的分布差异
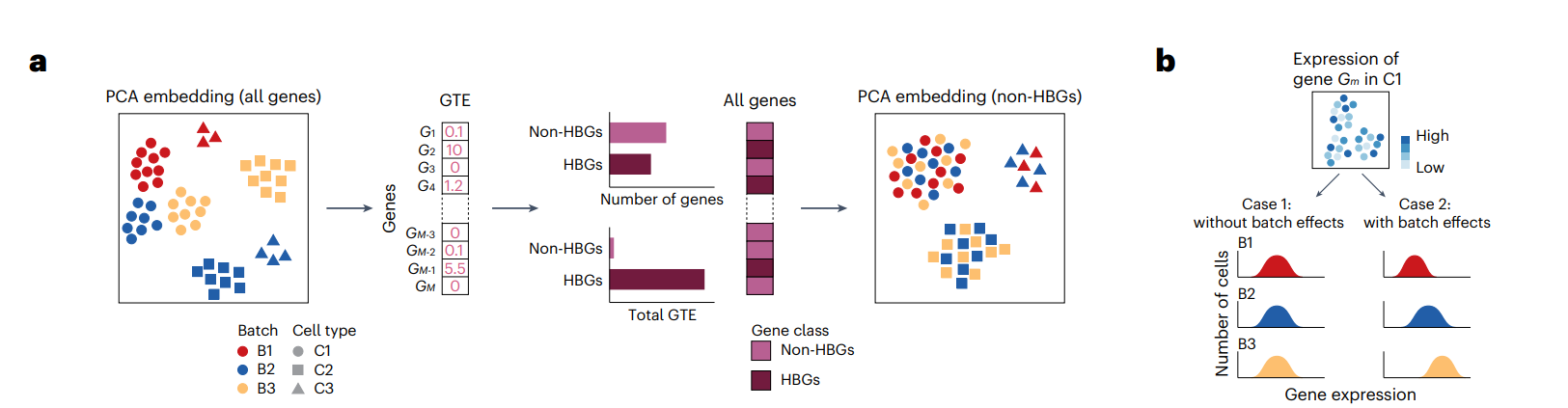
如上图所示,case2 中展示了一个在不同批次间差异分布的特征。分布差异的量化有多种方式,例如KL散度,组内-组间方差等。作者使用组内-组件方差来量化基因的批次差异。
给定基因$x \in R^{N} $, $N $是细胞数目,细胞有batch标签${b_1,b_2,…,b_L}$, 细胞类型标签(group)${g_1,g_2,…,g_K}$。有细胞类型变差
\[\begin{aligned} \delta_{group} &= \sum_{k=1}^{K} \sum_{n\in g_k}\frac{1}{N_k}(x_{n} - \mu_{k})^{2} \\ \mu_{k}&= \frac{1}{N_k}\sum_{n\in g_k}x_{n} \end{aligned}\]有批次-细胞类型变差
\[\begin{aligned} \delta_{batch-group} &= \sum_{l=1}^{L} \sum_{k=1}^{K} \frac{1}{N_k}\sum_{n\in g_k, n\in b_l} (x_n - \mu_{k,l})^{2} \\ \mu_{k,l}&= \frac{1}{N_{l,k}}\sum_{n\in g_k, n\in b_l}x_{n} \end{aligned}\]最终有GTE
\[\begin{aligned} &\delta = \delta_{group} - \delta_{batch-group} \\ & = \sum_{k=1}^{K} \frac{1}{N_k} \left [\sum_{n\in g_k}(x_n - \mu_k)^2 - \sum_{l=1}^{L} \sum_{n\in g_k, n\in b_l} (x_n - \mu_{k,l})^{2} \right] \end{aligned}\]中括号内第一项是基因在给定细胞类型下的全变差,第二项是基因在给定细胞类型各个批次下的全变差和。
如果没有批次效应,对给定的细胞类型k, 整体的均值应当和批次内均值类似,$\mu_k \sim \mu_{k,l}$, 因此有 $\delta \sim 0$。可以根据$\delta$的值来判断批次效应。
去除 HBG 可以有效较低批次效应
给定批次效应的量化后,是否在量化上批次无关的基因可以有效降低批次效应?我们将HVG中,对于批次敏感(GTE高)的基因定义为HBG(highly batch-sensitive gene)
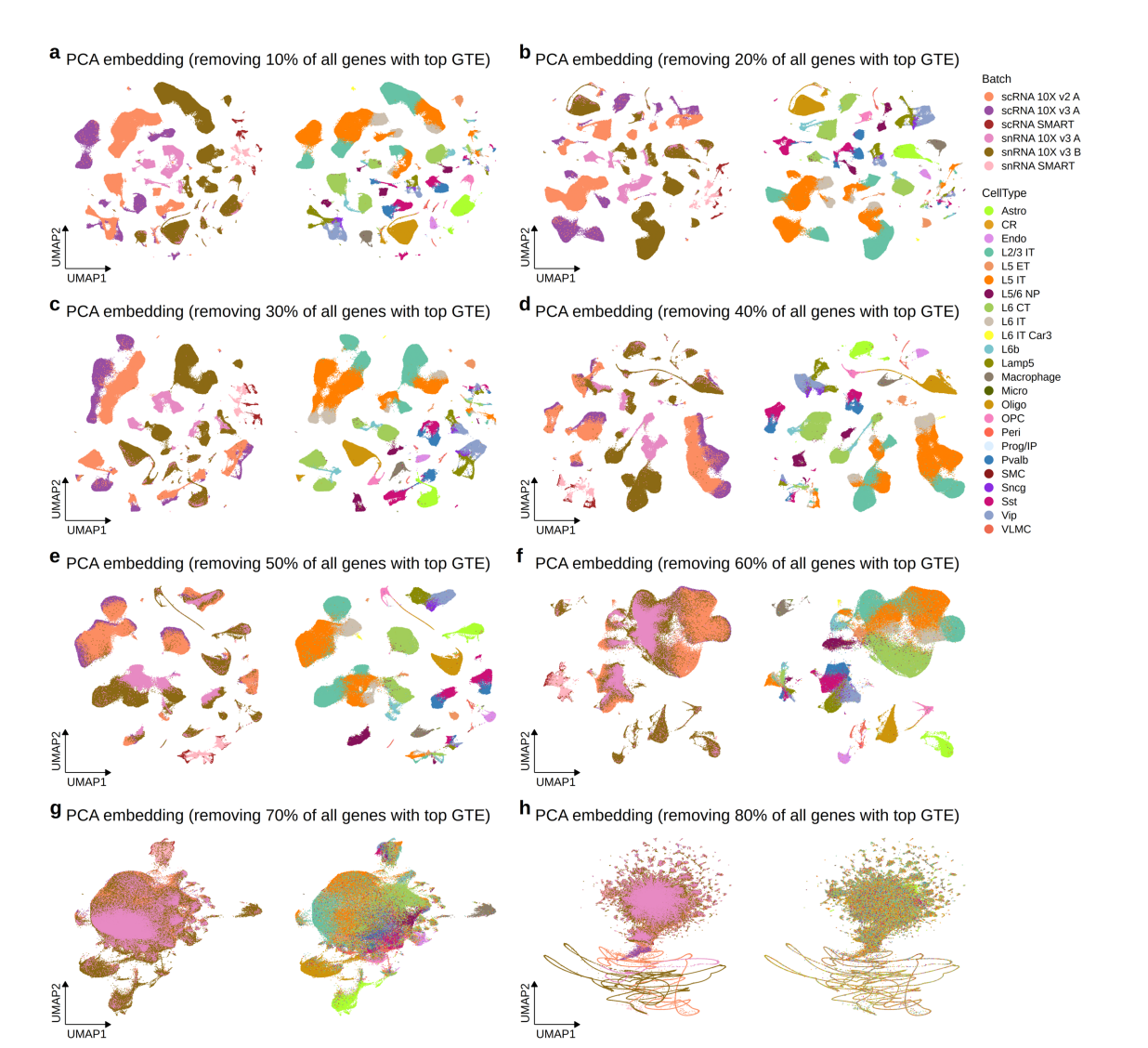
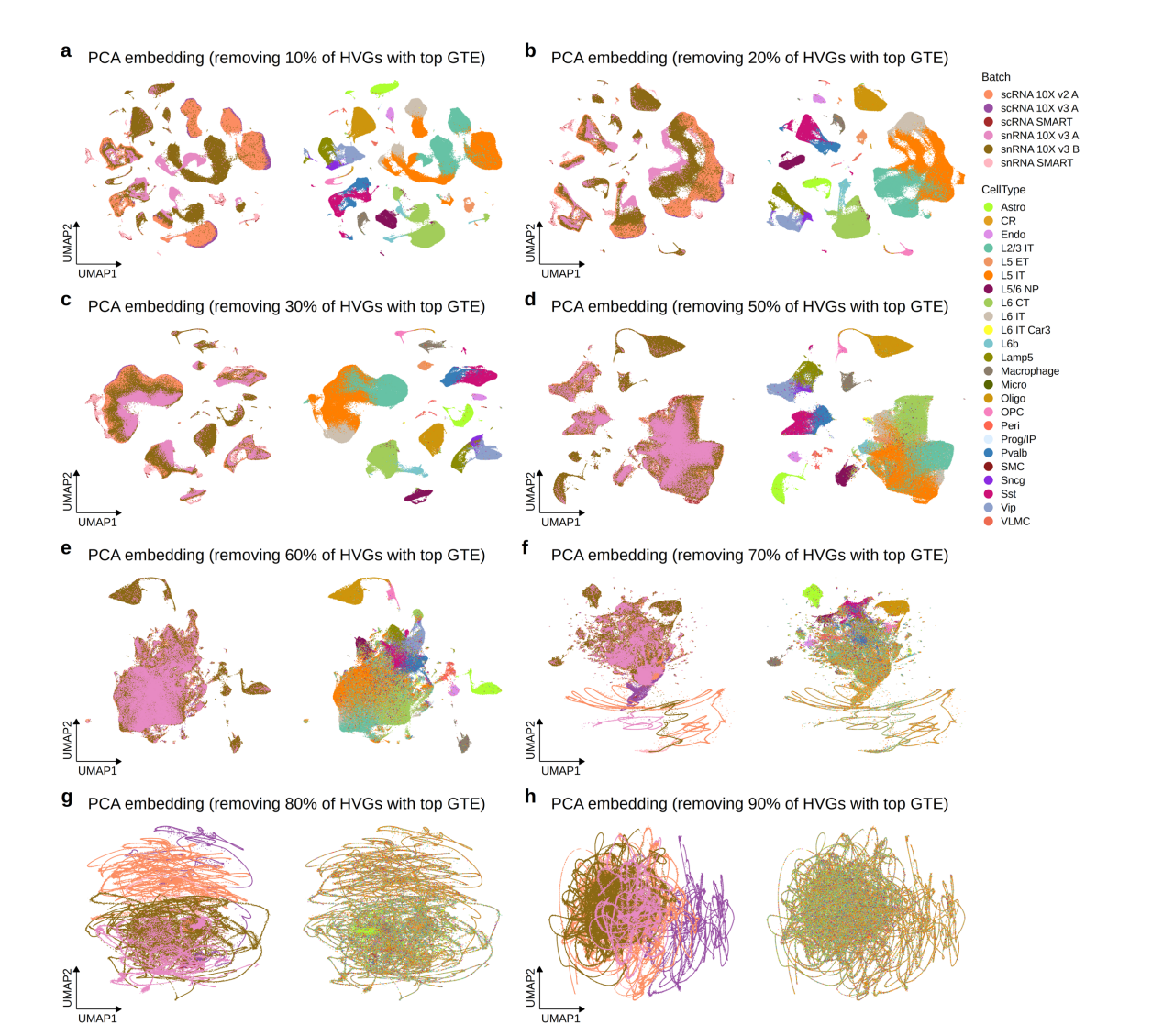
从下面的C,E图中可以看到, 在小鼠的初级运动皮层(MOP)中,共有6个batch的数据,可以看到有反应物版本 v2.v3, snRNA和scRNA, 10X和SMART-seq等批次差异,在使用4k hvg 直接 PCA+UMAP中,细胞类型没有一致性,但在去除HVG中GTE指标最高的40%基因后,批次效应有明显环节,除了L5 IT, L2/3 IT之外,基本都很好的对齐了,并且F图也验证了,这个效果的提升不是由于减少HVG数目带来的
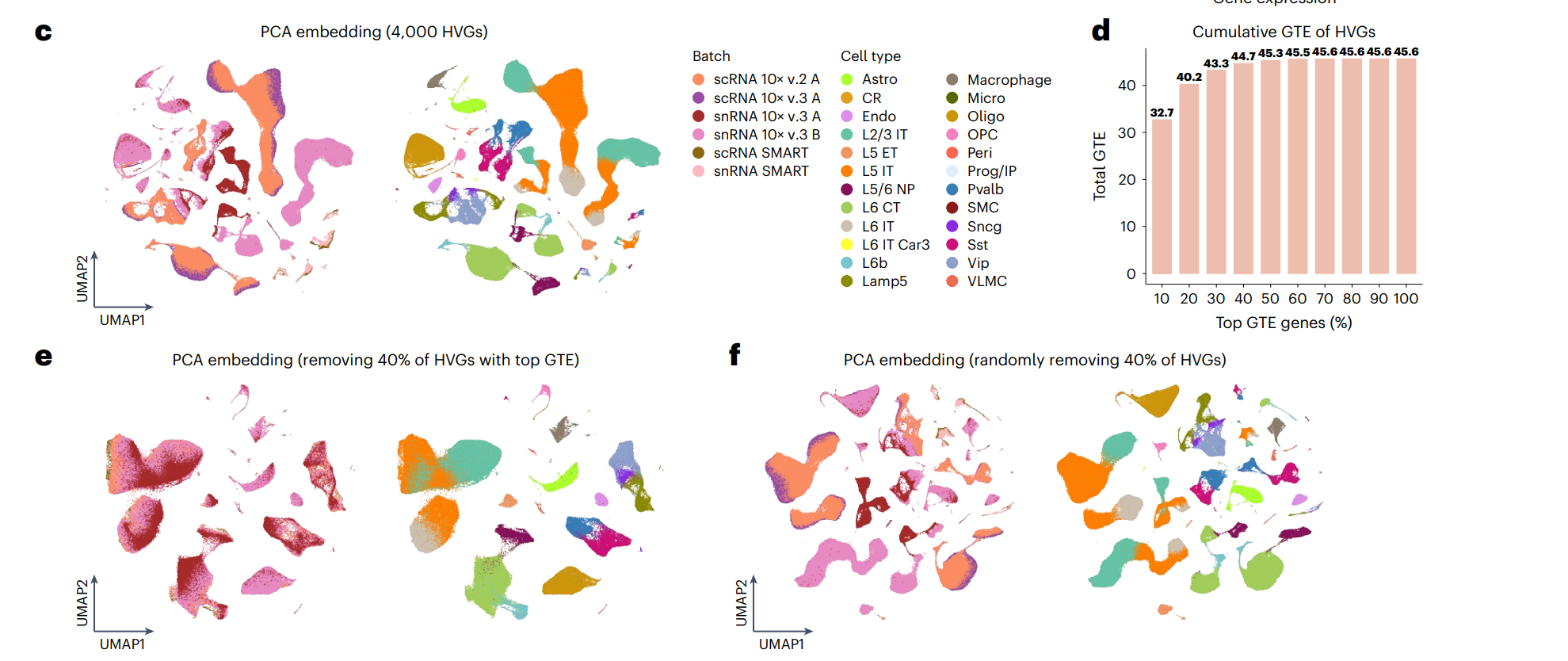
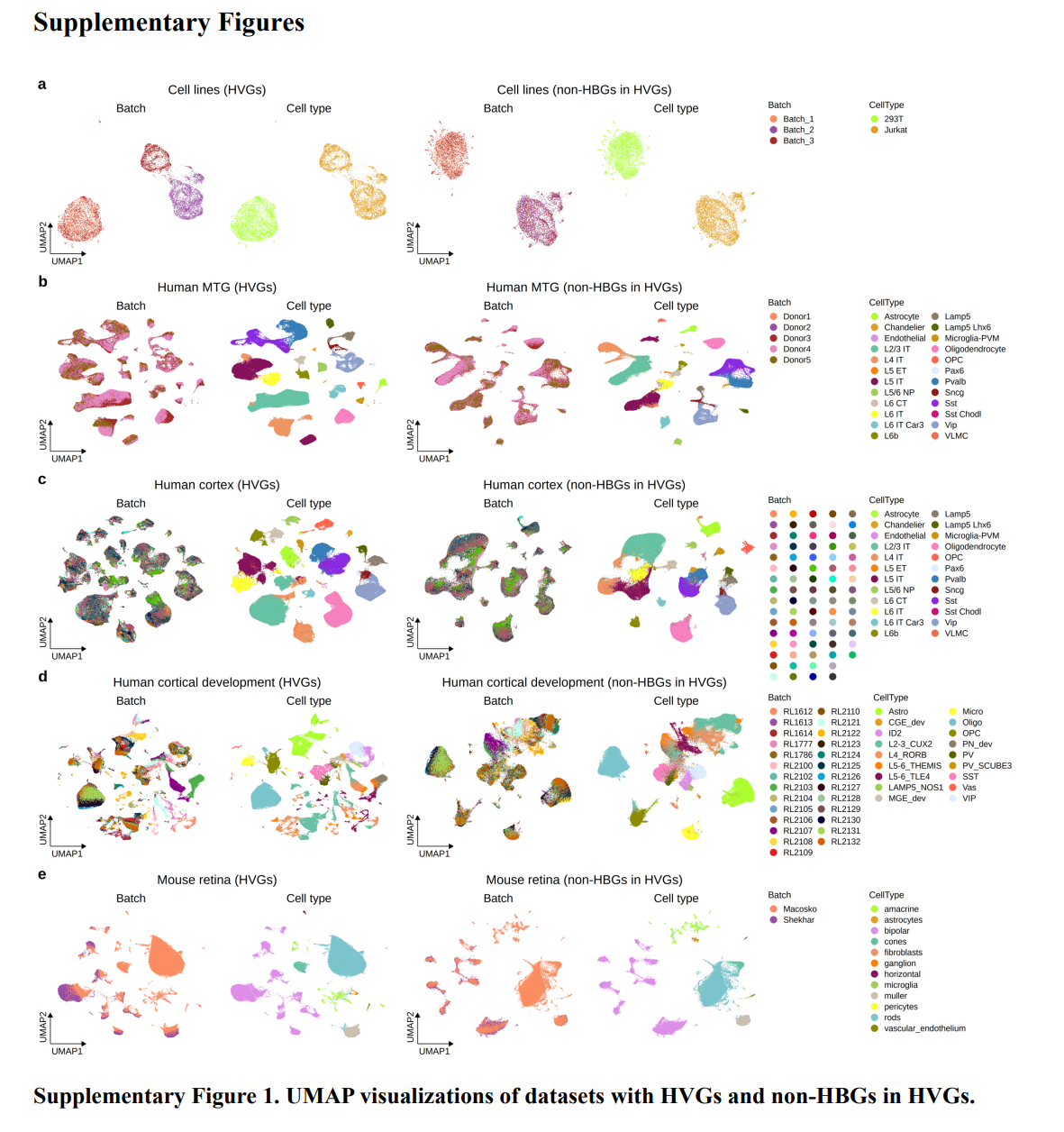
这个结果很棒,从附属材料来看,去除HBG的策略在绝大多数情况下都是奏效的。仅在Human MTG 和 cortex中,粉色的L4 IT 似乎没有被完全对齐。但这种差异也可能和原始的细胞注释有关。总的来说,non-HBG 可以有效较低批次效应。
HBG selection 算法如下:

注意这里的HBG 是定义在HVG中的,结合下图H可以看到,HVG的GTE远低于non-HVG的HBG。如果直接在全部基因中去除HBG,很有可能去除的都是non-HVG,在后续分析中依旧存在批次效应,如图6所示。图7是和图6的对比,在HVG中去除HBG,不同的HBG比例会对批次效应整合和细胞类型的可分性产生影响,需要选择一个合适的HBG比例。
另外一个有意思的结果是,mitochondrial genes(线粒体基因)和 ribosomal genes(核糖体基因)是HBG的比例很高。scanpy中是有过滤这两类基因的操作的,如果按照标准流程执行的话应当会自动的删除这些批次效应较强的基因。
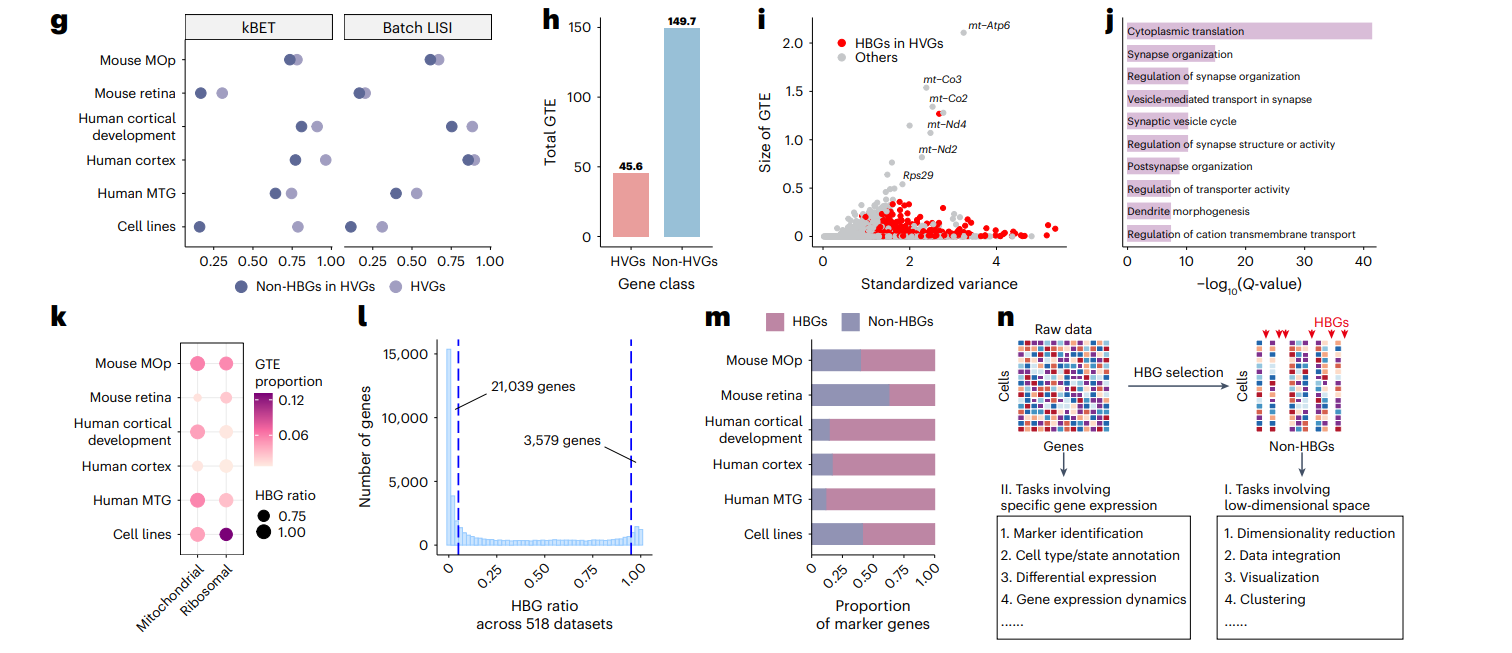
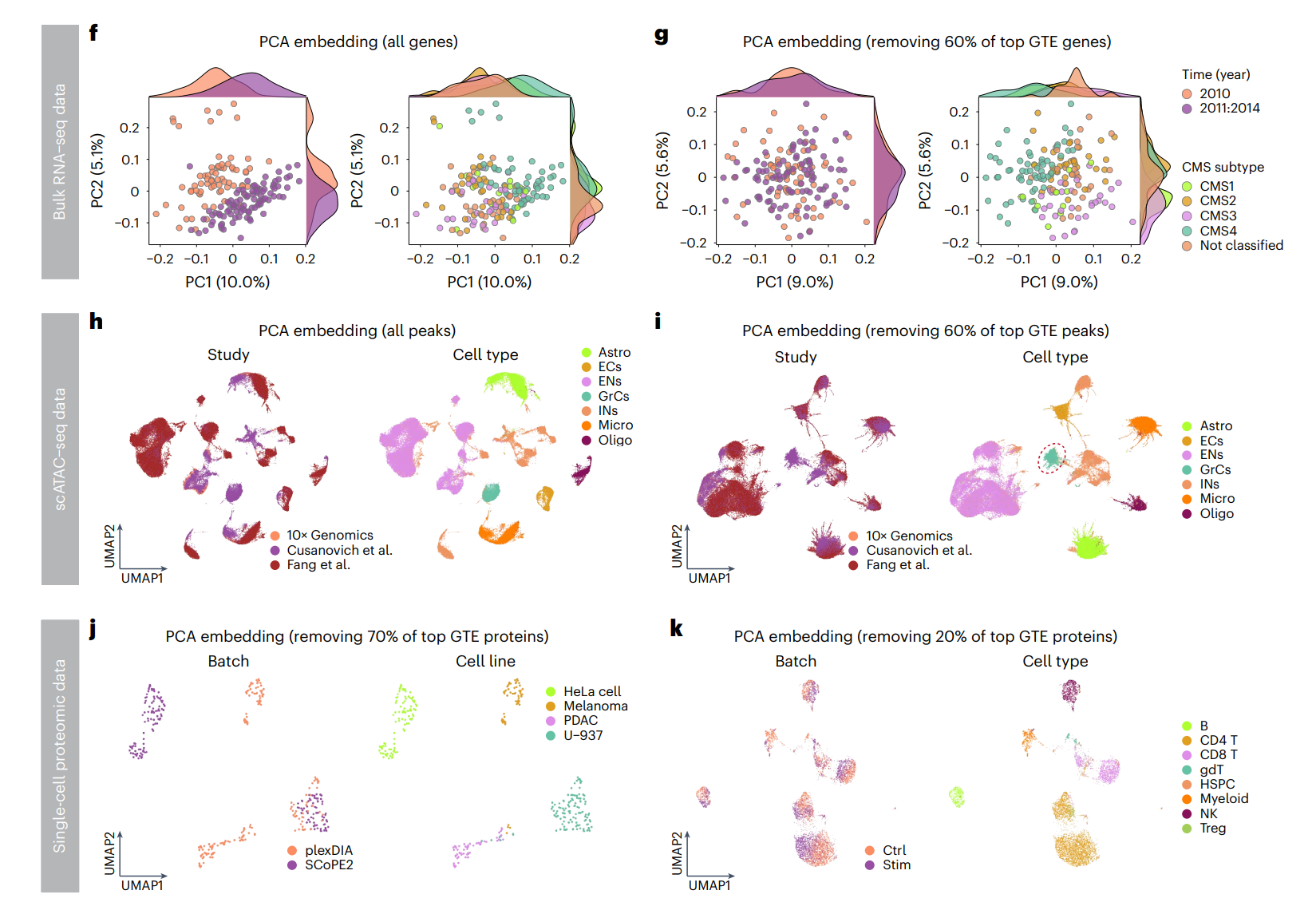
GTE 策略可以应用到其他组学上
下图展示了GTE 策略可以在其他组学数据上进行批次校正。
总结
GTE给出了一个简单有效的批次校正策略,同时也给出了一种数据整合算法的设计思路,可以从单个特征的分布校正的角度出发来设计。
GTE 存在一定局限性:
- 依赖注释信息。GTE需要 batch-label 和 group-label,其中group-label需要单个数据集去进行注释并统一不同数据集标签,这个要求对应用前景有一定限制性。假如单独的数据集都能准确注释了,直接把相同group拿出来差异分析就好了。一个可能的解决策略是在注释时使用coarse-label,之后应用GTE获得去除批次效应的cell-embedding再进行fine-label注释,查看是否会有差异细胞类型。
- 图5中子图m说明了一些HBG基因也是cell marker,因此去除HBG基因的粗略,存在细胞类型可分性和批次差异的一个平衡,在图7中也可以看到这个平衡和去除HBG比例的关联。